Um Moleküle anzusehen, benötigt man ein Elektronenmikroskop – ein solches ist jedoch recht teuer, oder eine passende Software. Unter Linux findet sich dazu unter anderem die Software RasMol – diese ist zwar auch für macOS und Windows verfügbar – aber nur unter Linux lassen sich auch zahlreiche Web-Tutorials anwenden. Wir sehen uns hier einmal diese Software etwas genauer an.
Inhaltsverzeichnis
RasMol installieren
Unter auf Debian basierenden Linux-Distributionen installieren Sie diese Software wie üblich ganz einfach über die Paket-Verwaltung durch das Paket „rasmol„, alternativ finden Sie diese Software auch unter RasMol.
RasMol nutzen
Sie finden diese Software nach der Installation im Anwendungsmenü unter der Kategorie Bildung, alternativ nutzen Sie den Schnellstarter (Alt+F2) oder das Terminal mit dem Befehl:
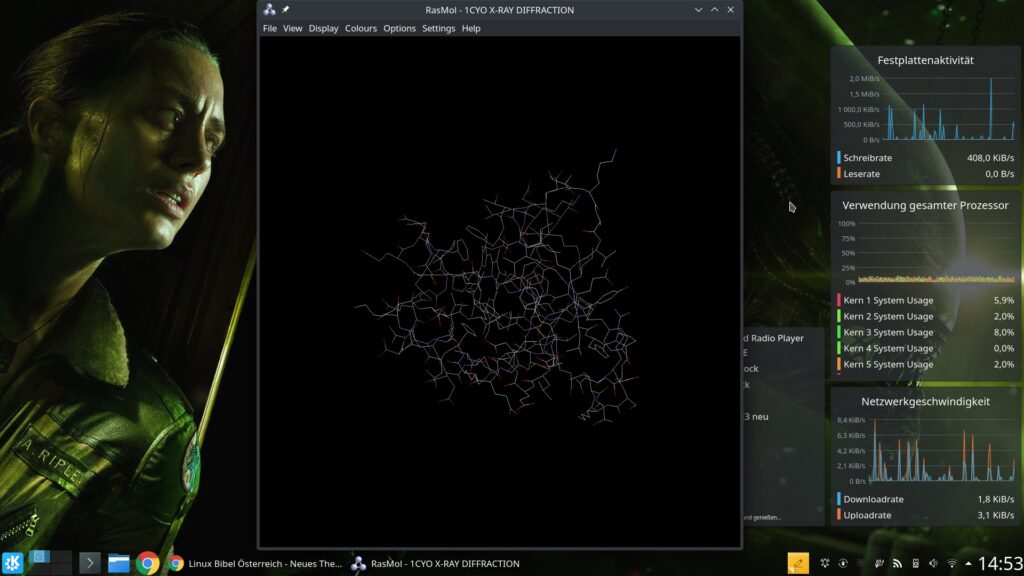
rasmol-gtkWas wir nun benötigen ist eine Beispiel-Datei für ein Molekül – hier akzeptiert die Software unter anderem Dateien im PDB-Format, ich nehme hier ein Beispiel aus der PDB-Datenbank für Proteine. In meinem Beispiel das Protein „3b5c“ und öffne dieses über das Menü „File → Open„:
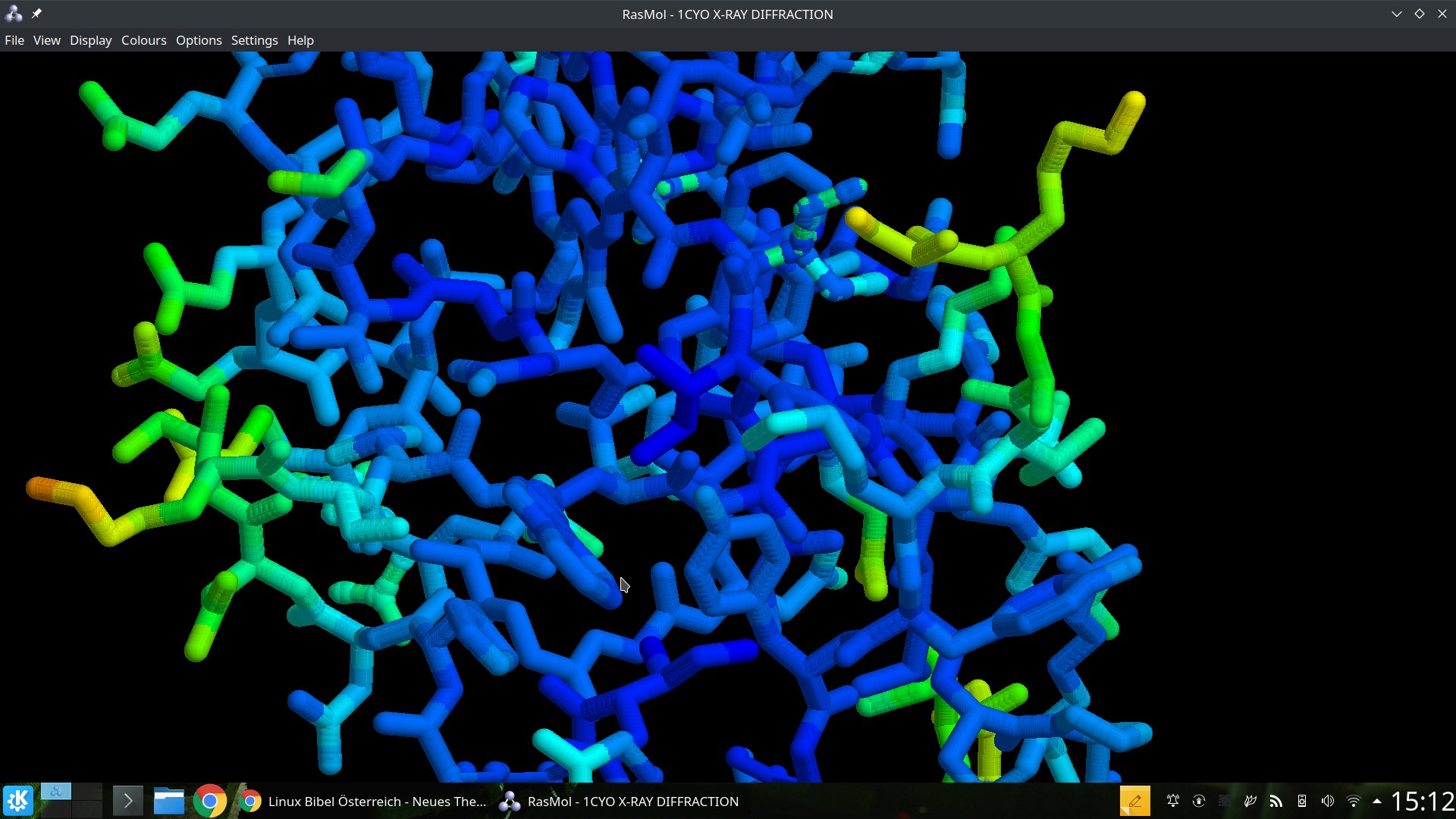
Über das Menü „Display“ schalten Sie zwischen verschiedenen Ansichten um, hier etwa „Sticks„:
Mit der linken Maustaste drehen Sie das Molekül, mit der rechten wird es verschoben. Mit den weiteren Menüs – etwa „Colors“ lässt sich die Ausgabe anpassen.
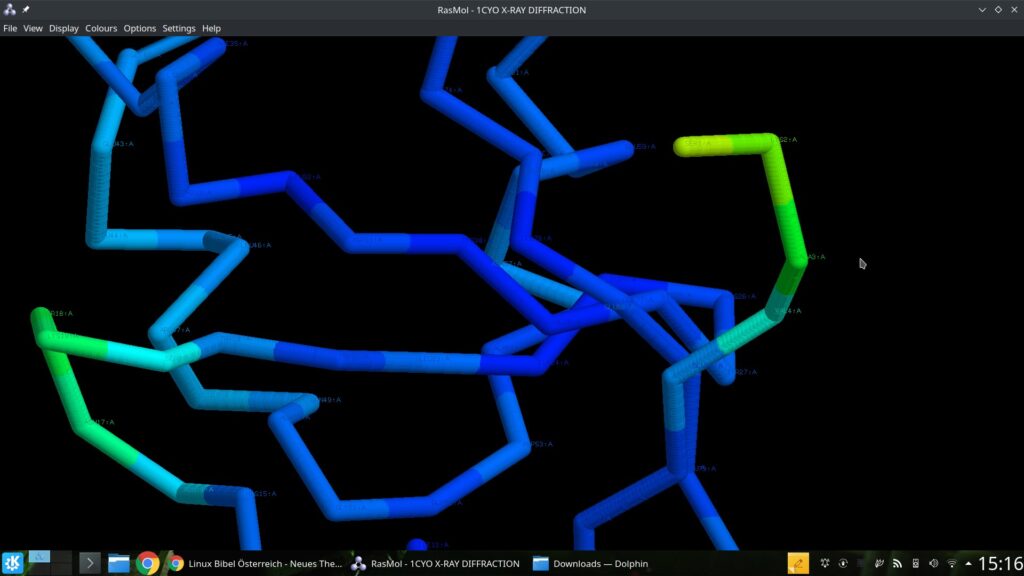
Mit Umschalt+Linksklick+Ziehen regeln Sie den Zoom:
Über das Menü „Options“ lassen sich wieder unter anderem die Bezeichnungen aktivieren und deaktivieren:
Die komplette Dokumentation der Software finden Sie unter RasMol Dokumentation.
Erstveröffentlichung: So 12. Dez 2021, 15:19




Noch keine Reaktion